RNA編輯:腫瘤和遺傳疾病治療的新選擇
2018年,美國FDA和歐洲EMA批準了來自RNA藥物制造兩大巨頭Ionis Pharmaceuticals公司的反義寡核苷酸療法藥物Tegesedi(Inotersen)和Alnylam Pharmaceuticals公司的siRNA療法藥物Onpattro(Patisiran),用于治療遺傳性轉運甲狀腺素蛋白淀粉樣變性,成為了RNA治療領域新的里程碑。近年來,RNA療法已經取得了較為長足的發展,大型制藥公司也正在不斷加大對于RNA藥物研究的投資。不過,傳統的RNA治療主要還是集中于ASO與siRNA兩大領域,而近年來呈研究上升趨勢的RNA編輯為RNA治療提供了新的選擇。那么本期的推送,小編就帶大家來看一看我們之前曾詳細分析過的RNA編輯是如何應用到腫瘤與遺傳疾病治療中!
正如小編剛剛總結的,傳統的靶向核酸的RNA治療是基于反義寡核苷酸(antisense oligonucleotides, ASO)和RNA干擾(RNAi)兩大原理[1]。它們主要都是通過與RNA分子進行互補而起到影響翻譯或降解RNA的效果,進而消除錯誤蛋白對細胞或機體的影響,一些相關藥物也已經獲批上市。此外,以CRISPR/Cas9為代表的DNA編輯技術已經發展相當成熟,在諸多遺傳病治療中已經顯示出其強大的能力,特別是它和腫瘤免疫治療相結合,已發展成為新一代基因編輯CAR-T技術。那么,在這樣的背景之下,為什么我們還要選擇RNA編輯作為治療方式呢?
一、傳統RNA療法與DNA療法面臨的挑戰
1. 傳統的RNA療法的局限
ASO與RNAi等傳統的RNA療法雖然取得了很多進展,但仍然存在以下問題:
-
較多藥物穩定性低,具有一定程度的脫靶性,且靶向藥物活性較低[2];
-
它們的脫靶往往會導致內源正常mRNA在翻譯前水平的非正常降解;
-
這些RNA 療法常常伴隨著一定程度的副作用,如ASO藥物常存在血液凝固異常,血小板減少以及腎臟損害等風險。
因此,傳統RNA療法的發展并非一帆風順。

圖1. 常見調節基因表達的反義機制[2]
2. DNA療法的局限
DNA療法(基因治療)在現有臨床上主要還是采用以病毒為遞送載體的過表達系統進行治療,而基因編輯治療整體上還是處在基礎研究向臨床前研究的過度階段。而此類治療方式也可能存在著以下問題:
-
蛋白分子量過大,使得通過病毒載體進行裝載及人體內遞送十分困難;
-
由于蛋白過表達可能引起潛在的脫靶效應;
-
外源蛋白表達有可能引起機體產生免疫反應;
-
機體內的抗體有可能會和蛋白發生中和反應從而導致編輯失敗。
正是因為存在著的種種小問題,疾病治療領域就更需要進行更多新的方法學嘗試,進而打開疾病治療的新大門。
二、RNA編輯的優勢
那么,從整體上看,RNA編輯的優勢究竟在哪里呢?讓我們還是回歸到基本的中心法則吧!
1. DNA編輯的顧慮
RNA的生物合成是依賴于DNA的。DNA作為大多數生物(除少數病毒外)遺傳信息的基本載體,在生命活動中扮演著重要的角色,但是這也決定了它必須要盡可能少地出現錯誤。雖然DNA編輯技術有著很強大的功能,但是對DNA進行編輯仍然在很多應用情景下存在著倫理和安全上的顧慮,這樣成為了DNA編輯在發展過程中的不可忽視的一大問題。
2. RNA編輯的獨到之處
相比于基因編輯是直接在DNA水平進行操作,RNA編輯則是通過ADAR等脫氨酶在RNA尺度上進行單堿基的校正,使得DNA水平的突變可以通過中介調整而產生正確的蛋白。這樣,我們就可以在完全不改變DNA序列的情況下糾正蛋白質水平的錯誤。此外,特定的RNA在體內存在的時間相對較短,相比通過編輯DNA來永久改變基因組,RNA編輯在安全性上也有著不言而喻的優勢。
編輯RNA為我們在翻譯前修復缺陷蛋白提供了新的思路和可能,是之前傳統的RNA療法和基因療法都無法做到的,有助于多種疾病的治療。
三、RNA編輯治療的進展
由于RNA編輯還是屬于較為新興的研究領域,所以還沒有真正進入臨床。但是在基礎研究領域,許多新的RNA編輯工具已經開始著眼于尋找疾病治療的應用,如單堿基遺傳疾病及癌癥等。1. 單堿基遺傳疾病治療
在迄今為止已知的諸多遺傳病中,致病類型最多的還是單堿基突變。研究統計結果表明,在已知的與疾病相關的50000種人類基因變異中,由于單堿基的改變而導致疾病的>30000種(圖2)。雖然這些突變可能會被ABE或CBE等基因編輯工具較正,但是基于ADAR等的“RNA單堿基編輯器”同樣可以在RNA水平糾正突變。下面是一些近期報道的RNA編輯治療遺傳疾病的成功案例。
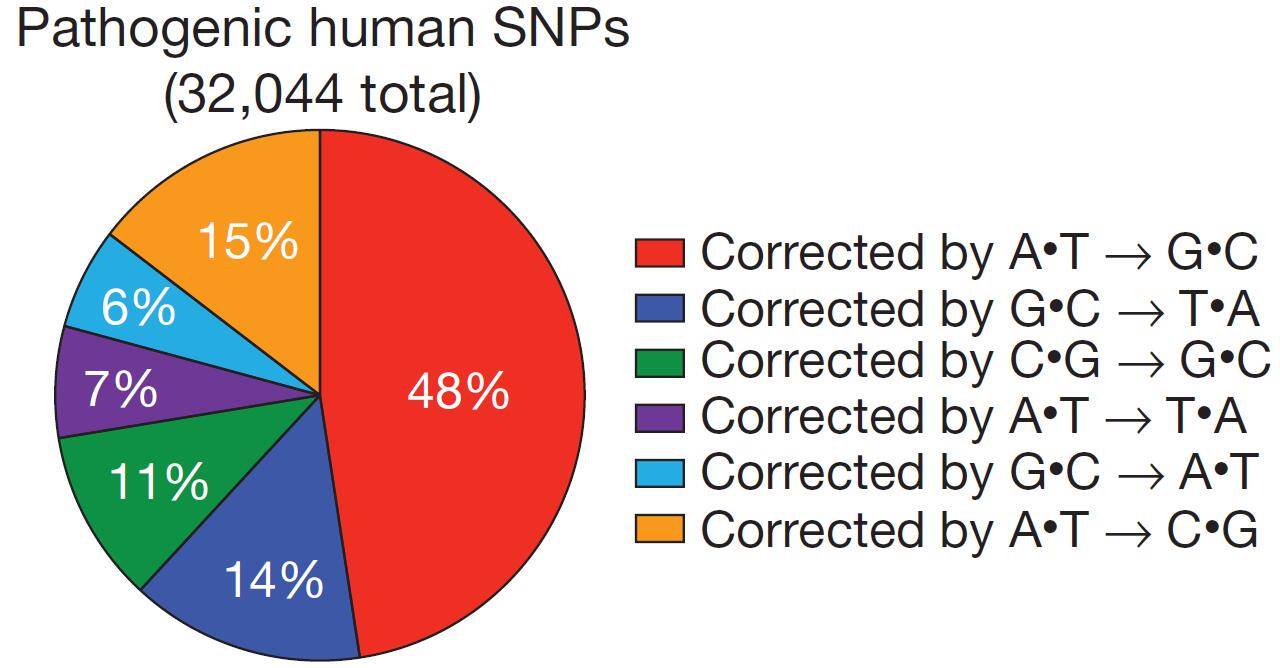
圖2. 人類遺傳病中單堿基突變類型
A1-AT綜合征α1-抗胰蛋白酶缺乏癥是血中抗蛋白酶成分:α1-抗胰蛋白酶(簡稱A1-AT)缺乏引起一種先天性代謝病。SERPINA1 (絲氨酸蛋白酶抑制劑家庭成員1)中的PiZZ突變(E342K)是A1-AT缺陷癥最主要的原因。由于PiZZ等位基因的缺失,抗胰蛋白酶功能喪失,對肺和肝臟造成嚴重損害。
來自德國杜賓根大學的Stafforst團隊今年1月在Nature Biotechnology提出的RSETORE工具(可見RNA編輯系列上一篇推文)通過程序化設計的ASO招募內源性ADAR成功修復了E342K的疾病突變,并且在細胞上可以達到接近30%的編輯效率[3]。

圖3. RESTORE修復A1-AT綜合征突變
Hurler綜合征
又稱IH型粘多糖病,是由于α-L-艾杜糖醛酸酶(IDUA)缺乏而導致的一種遺傳疾病。北京大學魏文勝課題組于年7月在Nature Biotechnology上提出的一種名為LEAPER的工具(可見RNA編輯系列上一篇推文),通過長鏈arRNA招募內源性ADAR對Hurler綜合征患者原代細胞中的致病突變進行了修復,且基本不出現脫靶現象[4]。

圖4. LEAPER對IDUA突變的校正
肌營養不良癥和鳥氨酸轉氨甲酰酶缺乏癥
這兩種疾病同樣也是來源于活性蛋白(或酶)的缺乏。加州大學圣地亞哥分校的Mali博士使用改造過的指導RNA,將ADAR結合并且將帶到正確RNA序列處進行編輯,在小鼠模型中修復了這兩類疾病的表型。這也是RNA編輯治療在動物體內成功的代表性工作[5]。

圖5. RNA編輯治療肌營養不良和鳥氨酸轉氨甲酰酶缺乏癥
2. 癌癥的治療如果說所有癌癥都有一個共同點的話,那就是無共同性。為什么每個人的腫瘤都不一樣?RNA編輯或許就是幕后黑手之一。腺苷(A)到肌苷(I)的RNA編輯給腫瘤轉錄組學中引入了許多核苷酸改變,造成腫瘤具有廣泛的個體異質性[6];而且異常RNA編輯使致瘤相關基因正常轉錄RNA翻譯出異常蛋白質,導致原蛋白質參與的各種生理功能的異常(如異常編輯PTEN或NF1等重要抑癌基因)。這就使得癌癥治療十分困難!
RNA編輯的出現可以對癥下藥,將出錯的腫瘤轉錄組或突變原癌/抑癌基因進行糾正,也可以對可能產生癌癥的潛在危害進行控制。
下面以抑癌基因P53和β-catenin為例,闡述RNA編輯如何對腫瘤相關基因進行糾正與調控!P53
抑癌基因TP53(P53)在維持細胞穩態中發揮著重要作用,在50%以上的人類癌癥中頻繁發生突變。TP53中c.158 G-to-A突變是一種臨床相關的無義突變(Trp53Ter),會導致非功能性的截短蛋白,進而誘發癌癥。同樣是剛剛所述的LEAPER工具能有效糾正突變導致的終止密碼子,進而產生正常的TP53蛋白[4]。

圖6. RNA編輯修復P53突變
β-cateninβ-連環蛋白(β-catenin)是一種與細胞正常生長相關聯的蛋白,它的磷酸化會導致蛋白的活化和細胞的生長。如果永久地發生這樣的改變,則細胞就會處于一種不受控制的生長狀態,可能導致癌癥的發生。
張峰團隊提出的RESCUE(RNA版本的CBE,見RNA編輯系列上一篇推文)工具通過對RNA的精準編輯,能直接激活β-catenin蛋白,促進細胞生長。如果使用的是傳統的DNA編輯手段,β-catenin蛋白就會被永久激活,導致細胞不受控的生長,甚至是產生癌癥。但利用RESCUE工具編輯RNA,只會促進短期的細胞生長。既可以促進傷口的愈合,也不會引起不可控的副作用。

圖7. RESCUE編輯β-catenin控制細胞生長
怎么樣,看過了小編的總結,是不是對RNA編輯在疾病治療里的應用前景充滿了期待呢?雖然現在RNA編輯在治療中的研究還是屬于初級階段,但是小編相信,隨著RNA研究浪潮的來臨,RNA編輯治療會和已經不斷前進發展的RNA療法主流融合并攜手,開創分子水平治療的新時代!
參考文獻
[1] Dowdy SF. Overcoming cellular barriers for RNA therapeutics. Nat Biotechnol. 2017, 35 (3):222-229.
[2] Crooke ST, Witztum JL, Bennett CF, et al. RNA-Targeted Therapeutics. Cell Metab. 2018, 27 (4):714-739.
[3] Merkle T, Merz S, Reautschnig P, et al. Precise RNA editing by recruiting endogenous ADARs with antisense oligonucleotides. Nat Biotechnol, 2019, 37 (2): 133-138.
[4] Qu L, Yi Z, Zhu S, et al. Programmable RNA editing by recruiting endogenous ADAR using engineered RNAs. Nat Biotechnol, 2019, 37 (9): 1059-1069.
[5] Katrekar D, Chen G, Meluzzi D, et al. In vivo RNA editing of point mutations via RNA-guided adenosine deaminases. Nat Methods, 2019, 16 (3): 239-242.
[6] Peng X, Xu X, Wang Y, et al. A-to-I RNA Editing Contributes to Proteomic Diversity in Cancer. Cancer Cell. 2018, 33 (5):817-828.
[7] Abudayyeh OO, Gootenberg JS, Franklin B, et al. A cytosine deaminase for programmable single-base RNA editing. Science, 2019, 365 (6451): 382-386.