基于雜交和分子標簽的新型錯誤矯正測序HYTEC-seq在臨床中的應用

循環腫瘤DNA(ctDNA)分析已成為一種重要的臨床有用的癌癥診斷和治療監測工具。然而,ctDNA檢測由于DNA濃度低,所以在技術上難度大且實驗情況復雜。這篇文章中,科學家們描述了其在Ion Torrent 測序平臺上新開發的一種高靈敏度的ctDNA檢測方法,被稱之為基于雜交和標簽技術的錯誤校正測序(HYTEC-seq)。該方法結合了雜交捕獲和分子標簽,以及新型variant caller PlasmaMutationDetector2以消除背景錯誤。科研家闡述了使用已知突變的對照樣本用HYTEC-seq檢測未知突變時,分析靈敏度高。此外,為了證明這種方法在臨床中的實用性,科學家分析了44例晚期胰腺癌患者的血漿樣本,發現57%的患者存在突變,等位基因頻率低至0.23%。文章發表在NATURE scientific report期刊。

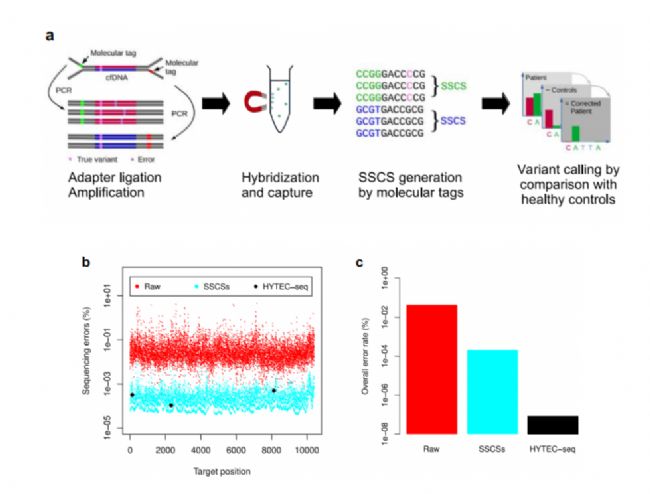
圖 1 (A)HYTEC-seq實驗流程 (B)原始測序reads、單鏈一致序列(SSCSs)和HYTEC-seq測序的每個目標位置的測序錯誤(替換)的相對百分比 (C)三種方法的總體錯誤率
實驗方法
· 樣本制備和cfDNA提取
整個實驗中,科學家選取了四個細胞系和60個健康人的樣本,也檢測了44個病人的血漿樣本。實驗采用50ng Multiplex I cfDNA Reference Standard Set(Horizon)作為標準品驗證HYTEC seq的可靠性。將患者和對照組的外周血收集到EDTA管中,并進行淋巴細胞梯度離心,將血漿在17000×g下離心10 min,去除細胞殘余和剩余血小板。采用QIAamp Circulating Nucleic Acid Kit (Qiagen)從血漿中提取cfDNA。提取后的cfDNA儲存在-80℃待用。
· HYTEC seq 文庫設計和構建
該文庫使用Y型接頭適用于Ion Torrent測序平臺,帶有Ion Torrent 平臺的A和P1接頭序列,樣本條形碼序列,和用于錯誤糾正的分子標簽。具體操作如下:
· 將20µL兩種新型的100µM P1和A接頭寡核苷酸序列在50µL的反應體積中分別退火
· 使用PCR儀將接頭寡核苷酸體系加熱到97.5°C,持續150s
· 關閉儀器,等待1小時讓接頭復性
· 將復性后的接頭根據cfDNA加入量用dd水稀釋待用
樣本DNA采取酶打斷法至160pb的片段,并使用磁珠進行富集。片段長度由安捷倫2100生物分析儀進行質控(High Sensitivity DNA kit)。文庫制備采用建庫試劑盒搭配安捷倫的靶向捕獲系統,采用了胰腺癌中常見突變的8個基因的靶向捕獲panel。捕獲到的DNA經PCR擴增后由磁珠富集。最終文庫由安捷倫2100生物分析儀質控。
在這篇測序方法開發的研究中,安捷倫2100生物分析儀又一次展現了其在核酸質控領域的強勢地位。科研家們使用的2100生物分析儀主要原理芯片式毛細管電泳結合微流控技術,操作簡單,檢測靈敏度高。在本次研究中老師選擇的High Sensitivity DNA kit,擁有極高的 DNA 分析靈敏度,適用于低至 5 pg/µL 的片段分析,或低至 100 pg/µL 的 NGS 文庫等復雜 DNA 樣品分析。文中提到的其他試劑耗材以及安捷倫2100生物分析儀配套現貨試劑詳見下表,各位老師走過路過不要錯過!